Rows: 153
Columns: 7
$ can <dbl> 1, 0, 1, 1, 0, 0, 1, 1, 1, 1, 0, 1, 1, 1, 1, 2, 2, 0, 1, …
$ can_intent <dbl> 1, 0, 0, 1, 0, 0, 0, 0, 4, 0, 0, 0, 2, 0, 0, 5, 4, 0, 1, …
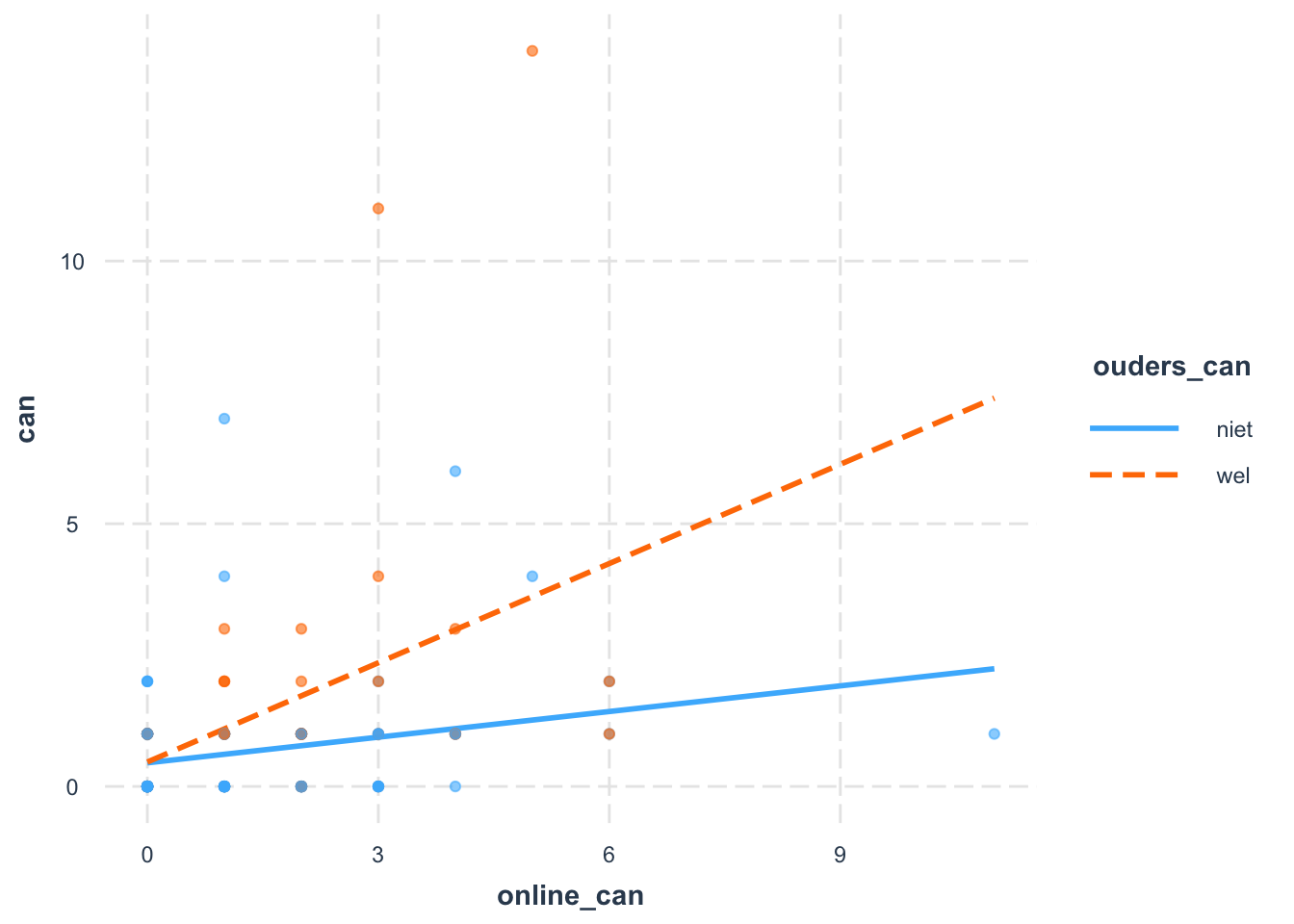
$ online_can <dbl> NA, 3, 1, 0, 0, 2, 2, 1, 4, 2, 0, 6, 2, 4, 0, 6, 1, 0, 4,…
$ ouders_can <chr> "niet", "wel", "niet", "niet", "niet", "niet", "wel", "ni…
$ sm_freq <dbl> 3, 4, 4, 4, 5, 6, 4, 5, 4, 3, 4, 3, 4, 4, 3, 4, 4, 4, 4, …
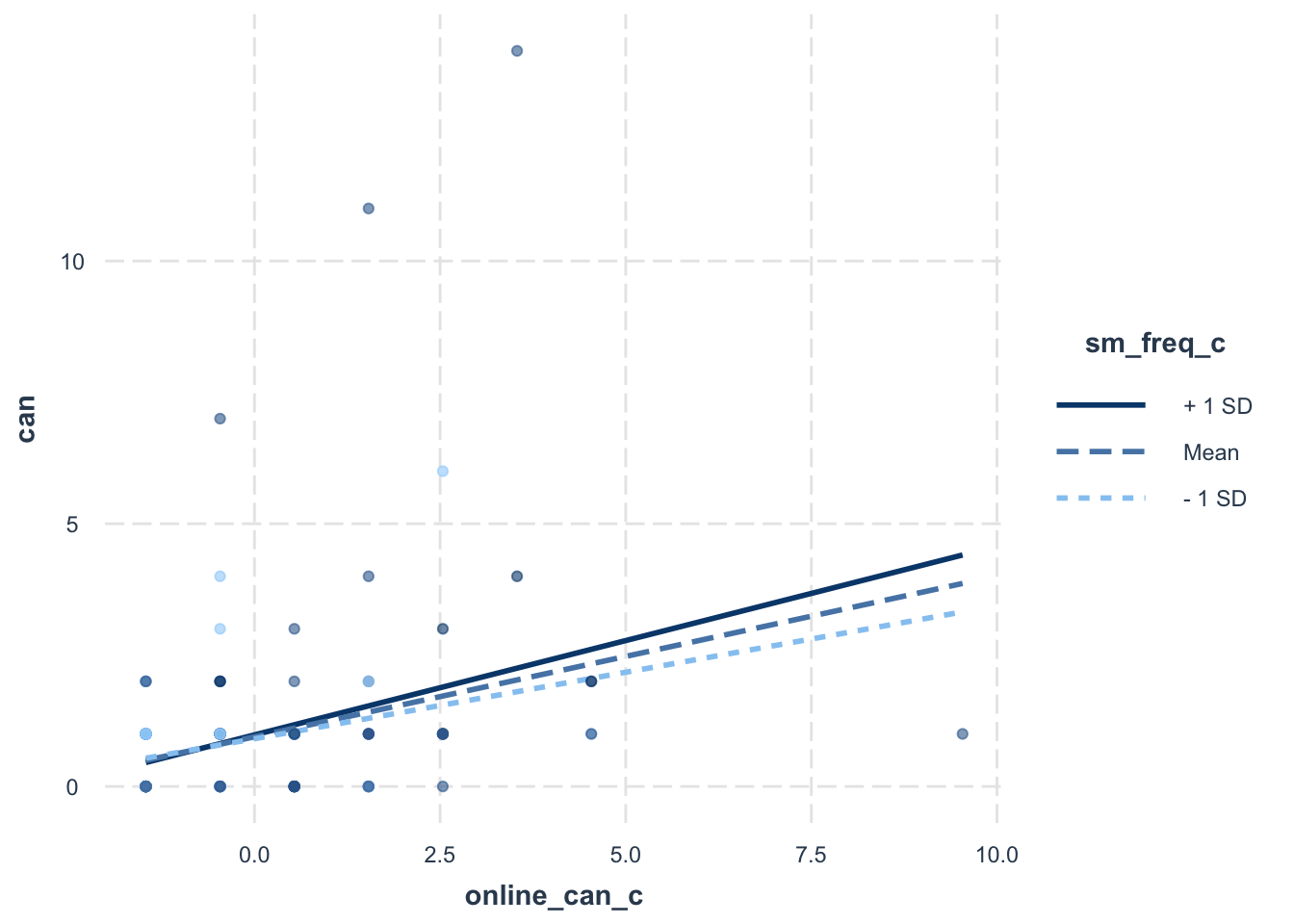
$ online_can_c <dbl> NA, 1.5367647, -0.4632353, -1.4632353, -1.4632353, 0.5367…
$ sm_freq_c <dbl> -0.7173913, 0.2826087, 0.2826087, 0.2826087, 1.2826087, 2…