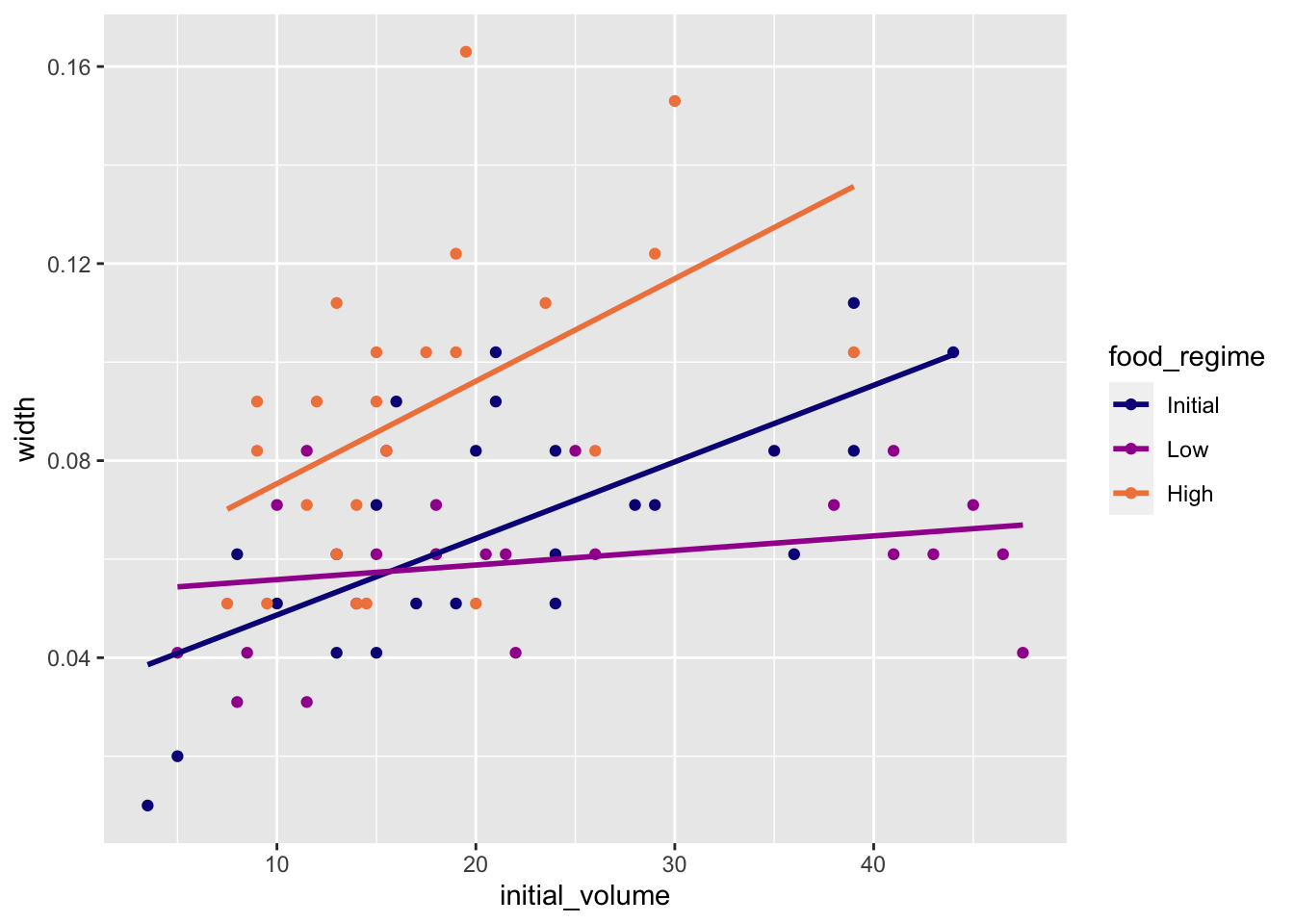
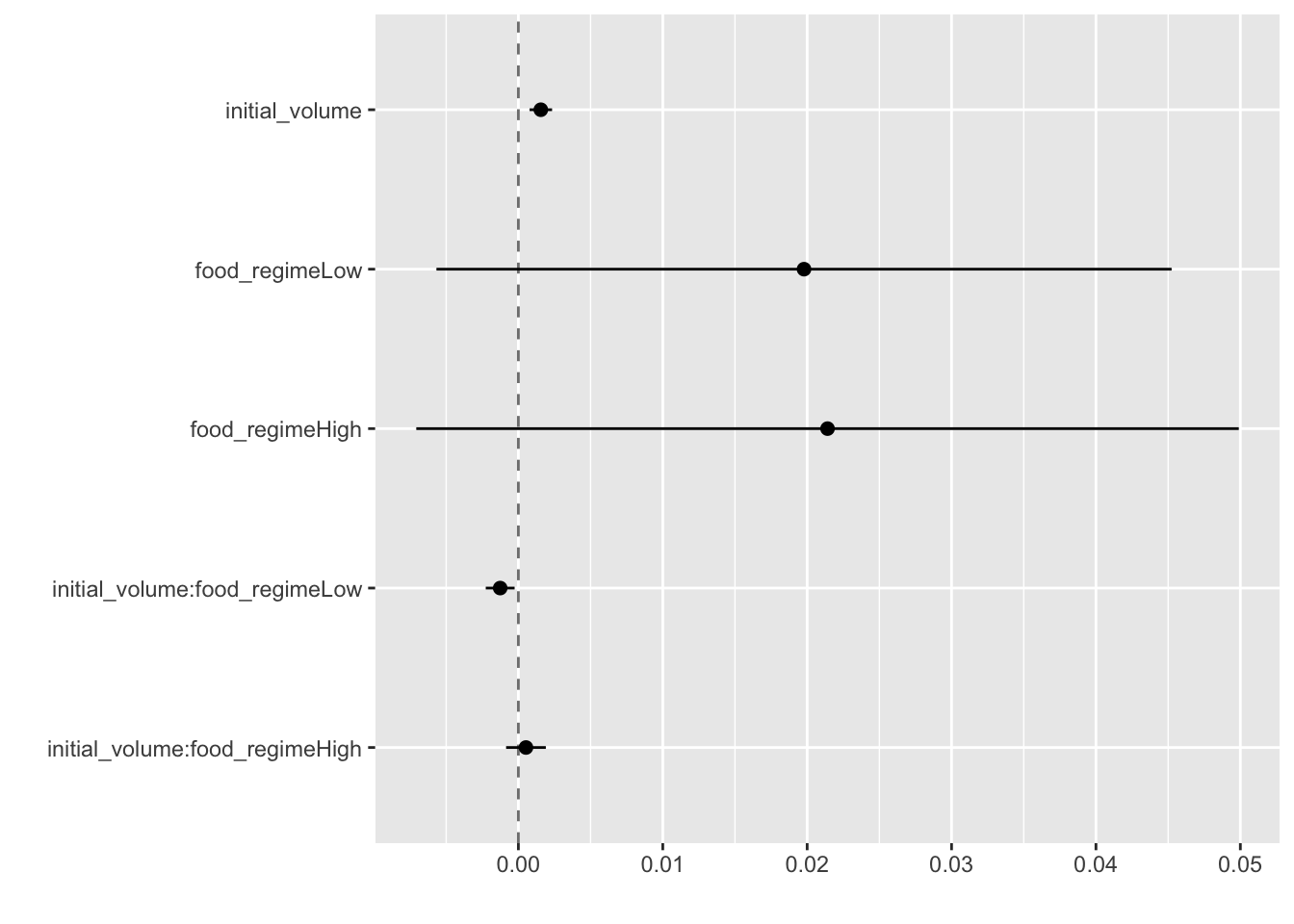
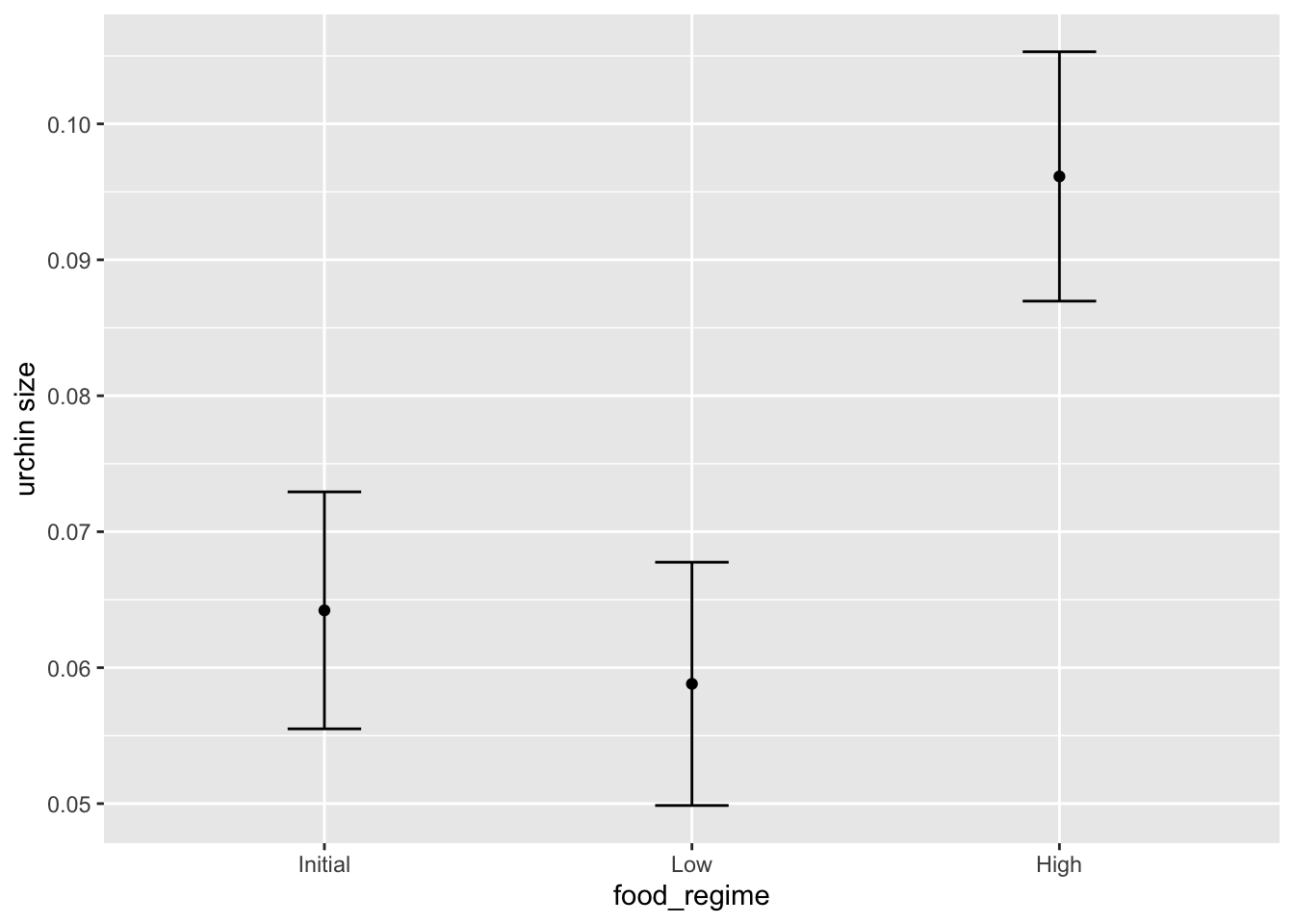
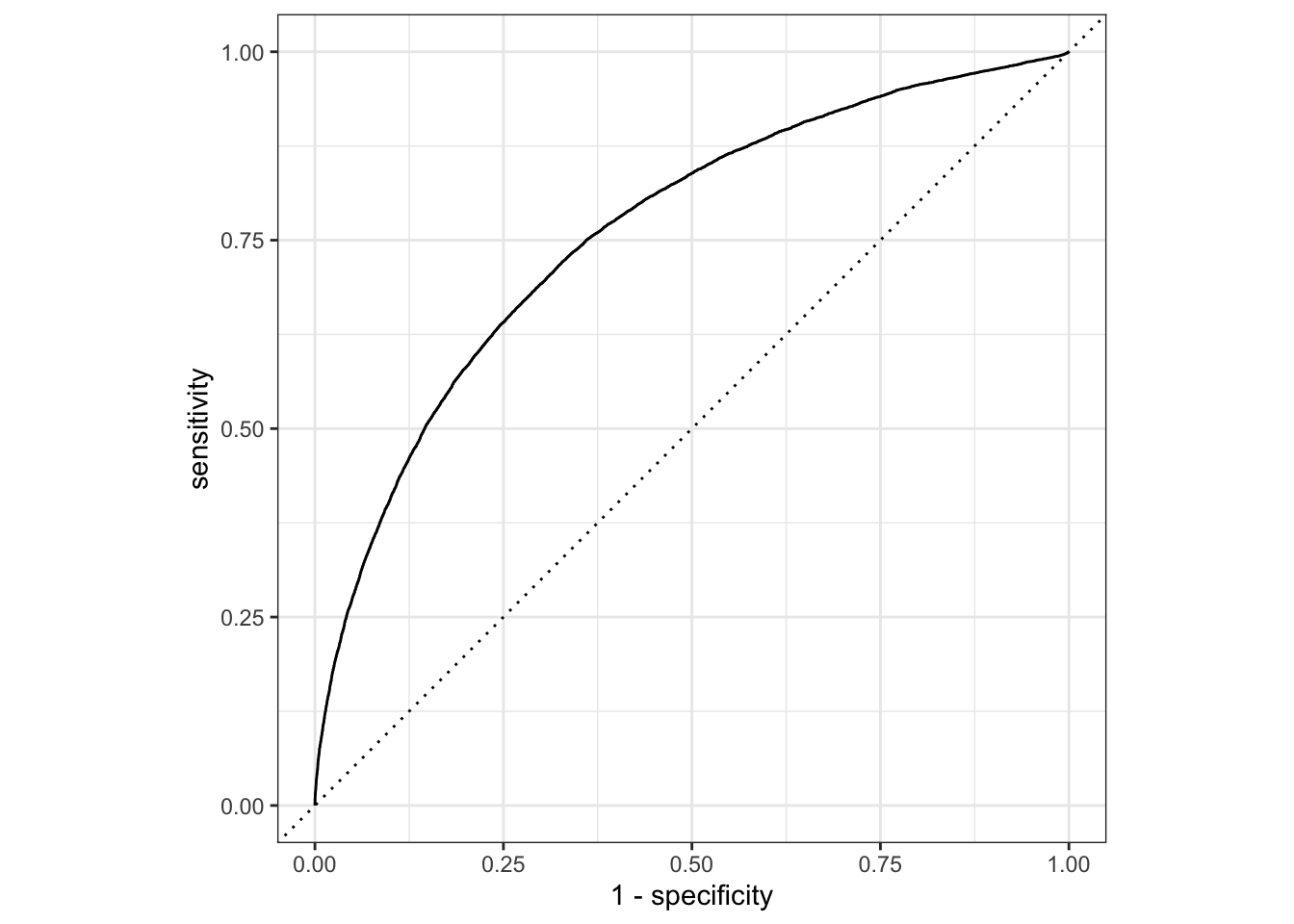
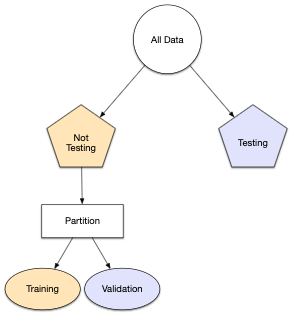
Rows: 50,000
Columns: 23
$ hotel <fct> City_Hotel, City_Hotel, Resort_Hotel, R…
$ lead_time <dbl> 217, 2, 95, 143, 136, 67, 47, 56, 80, 6…
$ stays_in_weekend_nights <dbl> 1, 0, 2, 2, 1, 2, 0, 0, 0, 2, 1, 0, 1, …
$ stays_in_week_nights <dbl> 3, 1, 5, 6, 4, 2, 2, 3, 4, 2, 2, 1, 2, …
$ adults <dbl> 2, 2, 2, 2, 2, 2, 2, 0, 2, 2, 2, 1, 2, …
$ children <fct> none, none, none, none, none, none, chi…
$ meal <fct> BB, BB, BB, HB, HB, SC, BB, BB, BB, BB,…
$ country <fct> DEU, PRT, GBR, ROU, PRT, GBR, ESP, ESP,…
$ market_segment <fct> Offline_TA/TO, Direct, Online_TA, Onlin…
$ distribution_channel <fct> TA/TO, Direct, TA/TO, TA/TO, Direct, TA…
$ is_repeated_guest <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
$ previous_cancellations <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
$ previous_bookings_not_canceled <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
$ reserved_room_type <fct> A, D, A, A, F, A, C, B, D, A, A, D, A, …
$ assigned_room_type <fct> A, K, A, A, F, A, C, A, D, A, D, D, A, …
$ booking_changes <dbl> 0, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
$ deposit_type <fct> No_Deposit, No_Deposit, No_Deposit, No_…
$ days_in_waiting_list <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
$ customer_type <fct> Transient-Party, Transient, Transient, …
$ average_daily_rate <dbl> 80.75, 170.00, 8.00, 81.00, 157.60, 49.…
$ required_car_parking_spaces <fct> none, none, none, none, none, none, non…
$ total_of_special_requests <dbl> 1, 3, 2, 1, 4, 1, 1, 1, 1, 1, 0, 1, 0, …
$ arrival_date <date> 2016-09-01, 2017-08-25, 2016-11-19, 20…